Nous avons mis au point des petits protocoles pour barcoder les espèces que l’on trouve sous terre. C’est à dire les identifier à partir d’une analyse de leur ADN.
Cette méthode procède en plusieurs étapes :
– prélever un échantillon et le mettre dans un tube avec de l’éthanol 95%
– extraire son ADN (protocole SDS / précipitation alcoolique)
– faire une amplification PCR d’une partie ciblé de son génome, COI (un petit bout de gène de la mitochondrie, présent chez tout les animaux)
– vérifier que l’amplification PCR a fonctionner en faisant une électrophorèse
– envoyer à séquencer dans une plateforme de séquençage
– Confronter la séquence de l’ADN (un fichier texte) avec une base de donnée en ligne.
les étapes d’extraction d’ADN / PCR / et électrophorèse se font avec un appareil tout en un, vendu par Bentolab : https://bento.bio/
Ils vendent également les pipettes et consommables associés à ces manip.
Extraction d’ADN



PCR : Réaction de polymérase en chaine
Où commander les primers (oligo) ?
https://www.eurogentec.com/en/custom-oligos
Thermoficher (en temps qu’association avec n° SIRET)
Elimbio
https://www.genewiz.com/en-GB/Public/Services/Oligo/
Quels primers choisir pour quels taxons …?
https://www.researchgate.net/figure/Relative-positions-of-primers-on-the-COI-barcode-region_fig1_281587817
Elbrecht V, Braukmann TWA, Ivanova NV, et al. Validation of COI metabarcoding primers for terrestrial arthropods. PeerJ. 2019;7:e7745. Published 2019 Oct 7. doi:10.7717/peerj.7745
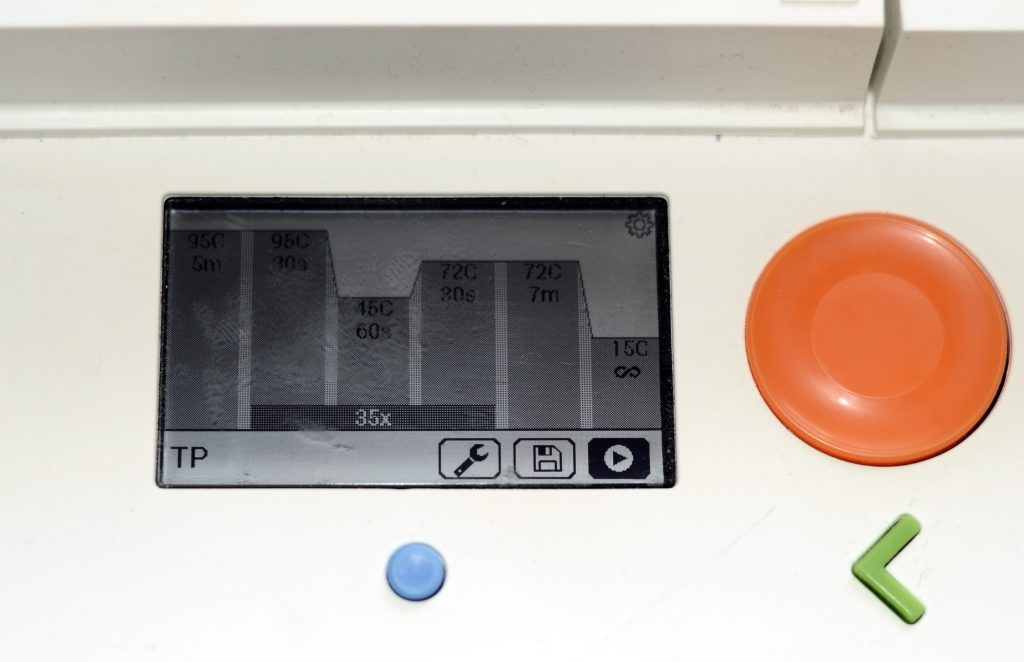
Quel programme : la Température d’hybridation (Tm) peut etre modifié en fonction des programmes entre 40 et 50°C.
Folmer :
(95°C 1min
tm = 40°C 1min
72°C 30sec)x35
72°C 5min

Electrophorèse


Séquences
Où envoyer à séquencer :
https://www.genoscreen.fr/fr/
Base de donnée de barcoding
NCBI Blast
http://www.boldsystems.org/