Petit bilan de fin d’année pour le projet de barcoding des specimens souterrain.
Nous avons uniquement testé le barcoding sur un gène mitochondriale COI (Cytochrome Oxidase I), et cela pour plusieurs échantillons collectés dans les souterrains francilien. Ce gène code pour une protéine impliquée dans la chaîne de transport des électrons dans la mitochondrie, une étape clé de la respiration cellulaire. Il est assez conservé entre différentes espèces pour exister chez « tout le monde » avec une base commune qui permet de designer des oligos pour l’amplifier, et assez variable pour avoir quelques modifications sur des changements de nucléotides ponctuels pour pouvoir identifier les espèces.
La mise au point de protocole consiste à tester plusieurs couples d’oligos pour la PCR par espèce supposées, sachant qu’il peut y avoir un mismatch du à une mutation sur la séquence cible. La biblio nous oriente vers des choix d’oligos privilégiés.
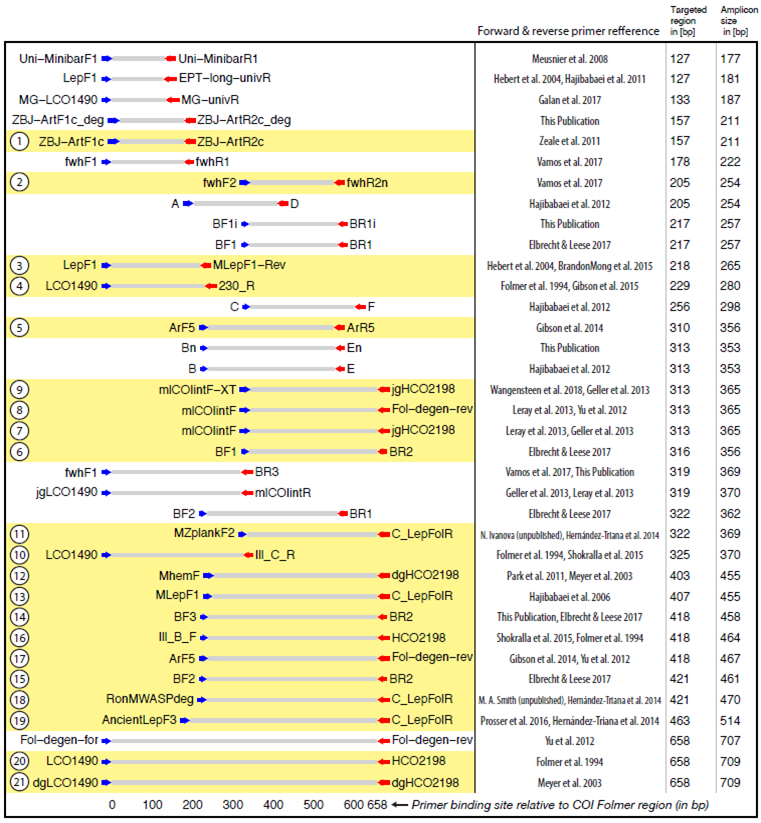
J’ai ensuite constitué mon set d’oligos à partir des données de séquences déjà connues par rapport aux identifications présumées, en vérifiant les sites les plus polymorphes chez celles ci pour que ca impacte le moins possible l’appariement spécifique oligos/ADN matrice.
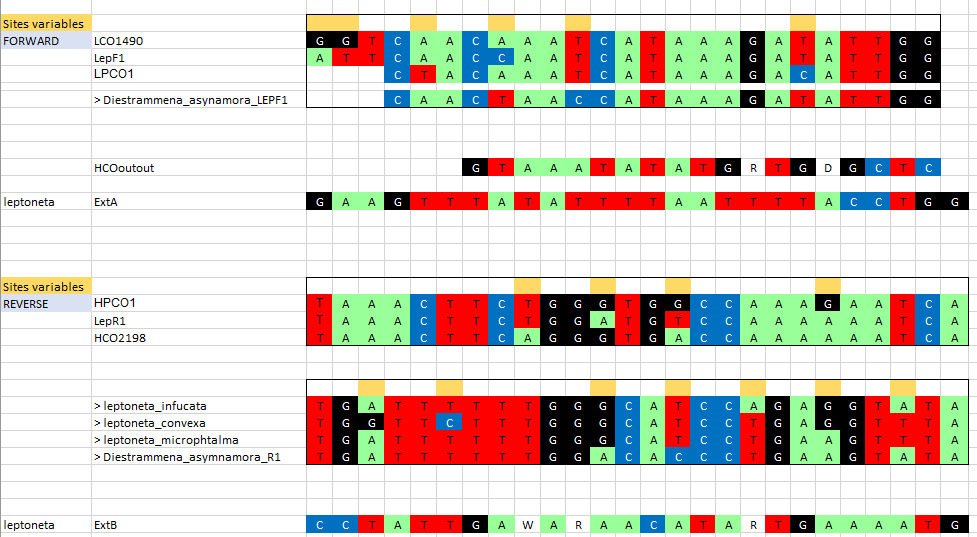
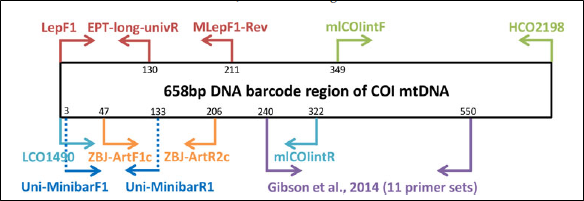

La mise au point de protocole se fait également sur le cycle des PCR, l’ajustement de la température de melting principalement.
J’ai retenu ce cycle le plus souvent efficace :
| good | |
| 95°C 2min | |
| 75°C 1min |
Sur les sessions d’envoi de 2020 voici les identifications obtenues :
| taxons attendus | Blast 1er match | Max score | query cover | E value | % ident | a priori blast | PCR OK | cycle ok | |
| Eupodes sp. | Eupodes sp. | 538 | 80% | 0,00E+00 | 85,00% | arachnid | 2021 | LCO1490+HCO2198 | 55^30_40c |
| Limonia nubeculosa | Limonia nubeculosa | 933 | 92% | 0 | 92,25% | 2020 | LCO1490+HCO2198 | 55^30_40c | |
| Collemboles d elevage des géants | Coecobrya tenebricosa | 1206 | 87% | 0.0 | 99.85% | collembola | 2021 | LCO1490+HCO2198 | 50°C 40cy |
| Nesticidae | Kryptonesticus eremita | 1173 | 94% | 0.0 | 99.53% | arachnid | LCO1490+HCO2198 | 50°C 40cy | |
| Nesticus | Kryptonesticus eremita | 1184 | 94% | 0.0 | 99.84% | arachnid | LCO1490+HCO2198 | 50°C 40cy | |
| Nesticus | Atypus jianfengensis | 420 | 99% | 6,00E-119 | 77.91% | arachnid | LCO1490+HCO2198 | 50°C 40cy | |
| Scotolemon sp. | Stygnus polyacanthus | 320 | 82% | 2,00E-87 | 77.29% | opiliones | LCO1490 + lepR1 | 50°C 40cy | |
| Scotolemon sp. | Stygnus polyacanthus | 322 | 81% | 7,00E-88 | 77.26% | opiliones | LCO1490 + lepR1 | 50°C 40cy | |
| Scotolemon sp. | Stygnus polyacanthus | 309 | 81% | 5E-84 | 76.82% | opiliones | LCO1490 + LEPR1 | 50°C 40cy | |
| Scotolemon doriae (Pavesi, 1878) | Haplodrassus umbratilis | 344 | 92% | 1,00E-92 | 76,60% | arachnid | LepR1 + LepF1 | 45°C 40cy | |
| Scotolemon doriae (Pavesi, 1878) | Stygnus polyacanthus | 235 | 87% | 6,00E-62 | 76.46% | arachnid | LCO1490 + LEPR1 | 45°C 40cy | |
| Nelima sp. | Mitopus glacialis | 363 | 83% | 4,00E-100 | 77,46% | opiliones | 2021 | LCO1490+HCO2198 | 55^30_40c |
| leptoneta olivacea | Leptoneta paroculus | 540 | 65% | 7,00E-156 | 88,11% | leptoneta | LCO1490 + LEPR1 | 50°C 40cy | |
| leptoneta olivacea | Leptoneta paroculus | 542 | 62% | 2,00E-156 | 88.11% | leptoneta | LCO1490 + LEPR1 | 50°C 40cy | |
| leptoneta olivacea | Mecysmauchenius sp. | 573 | 91% | 4,00E-159 | 84,26% | arachnid | 2021 | LepF1-R1 | 45°C 40cy |
| leptoneta abeilliei ? | Leptoneta infuscata | 756 | 97% | 0.0 | 87.11% | leptoneta | LCO1490 + lepR1 | 50°C 40cy | |
| leptoneta abeilliei ? | Leptoneta infuscata | 761 | 97% | 0.0 | 87.07% | arachnid | LCO1490 + lepR1 | 50°C 40cy | |
| leptoneta abeilliei | rien | leptoneta | LCO1490 + lepR1 | 50°C 40cy | |||||
| leptoneta abeilliei | Diplocentrus diablo | 158 | 79% | 2E-36 | 72.28% | arachnid | LCO1490 + lepR1 | 50°C 40cy |
Comme les banques de données sont alimentées depuis 2020, j’ai tenté de re blaster ces séquences. Les leptoneta sont bien toute des l. olivacea pour le moment parmi celles testées.
| n° BC | taxons attendus | Blast 1er match | Max score | % ident |
| reblast 2020 en 2024 | ||||
| BC030 | leptoneta olivacea | leptoneta olivacea | Bolt id | 98.57% |
| BC034 | leptoneta olivacea | leptoneta olivacea | Bolt id | 98.26% |
| BC025 | leptoneta olivacea | leptoneta olivacea | Bolt id | 99,22% |
| BC041 | leptoneta abeilliei ? | leptoneta olivacea | Bolt id | 98.57% |
| BC042 | leptoneta abeilliei ? | leptoneta olivacea | Bolt id | 87.07% |
Pour les échantillons de 2024 :
| n° BC | taxons attendus | Blast 1er match | Max score | query cover | E value | % ident | a priori blast | PCR OK | cycle ok | |
| BC058 | Leptoneta femelle | Leptoneta olivacea | Bolt id | 98.89% | 2024 | LCO1490 + lepR1 | 50°C 40cy | |||
| BC034 | leptoneta olivacea | Leptoneta olivacea | Bolt id | 98.57% | 2024 | |||||
| BC033 | Heteromurus nitidus ? | Heteromurus nitidus | Bolt id | 100% | 2024 | LepF1-R1 | 50 60sec | |||
| BC063 | collemboles de pixel 2 | Heteromurus nitidus | 717 | 98 | 0 | 87,66 | collembola | 2024 | LCO1490 + lepR1 | |
| BC031 | leptoneta olivacea femelle | Leprolochus birabeni | 662 | 98 | 0% | 85% | arachnid | 2024 | LepF1-R1 | 50 60sec |
| BC057 | Leptoneta olivacea male | Leprolochus birabeni | 688 | 96 | 0% | 85,08% | arachnid | 2024 | LCO1490 + lepR1 | 45°C 40cy |
| BC055 | Leptoneta male | Leptoneta infuscata | 769 | 95 | 0% | 87,15% | arachnid | 2024 | LCO1490 + lepR1 | 50°C 40cy |
| BC072 | Leptoneta sp | Leptoneta infuscata | 761 | 97 | 0 | 87,07% | arachnid | 2024 | LepF1-R1 | 50 60sec |
| kk1 | guano pûiselet | Philodina sp. | 243 | 98 | 2,00E-59 | 92.17% | – | 2024 | HCO3298 LCO1490 | 50 60sec |
Conclusion : Il n’est pas facile de faire confiance en les identifications par barcoding sans coupler cela à une identification morphologique. Concernant les espèces comme l. abeilliei ou Scotolemon doreae, il n’y a pas encore de séquence dans la base de données, c’est donc impossible d’avoir l’identification attendue. Pour certains échantillons juvéniles ou abimés du genre Kryptonesticus, cela fonctionne pas mal. Les identifications Limonia nubeculosa, Heteromurus nitidus et Coecobrya tenebricosa sont confirmé par un diagnostic sur phénotype.
Une curiosité à vérifier est la présence de Philodina sp. dans le guano de chauve-souris, et l’acarien du genre Eupode sp. Etant donné qu’il y a peu de spécialistes de ces groupes, c’est une information intéressante et nouvelle pour notre inventaire, mais à confirmer.
